双脱氧(确定核酸中核苷酸序列的方法)

双脱氧又名Sanger双脱氧链终止法,是用以确定核酸中核苷酸序列的一个方法。Sanger法是根据核苷酸在某一固定的点开始,随机在某一个特定的碱基处终止,并且在每个碱基后面进行荧光标记,产生以A、T、C、G结束的四组不同长度的一系列核苷酸,然后在尿素变性的PAGE胶上电泳进行检测,从而获得可见DNA碱基序列的一种方法。
双脱氧
生物
双脱氧测序
简介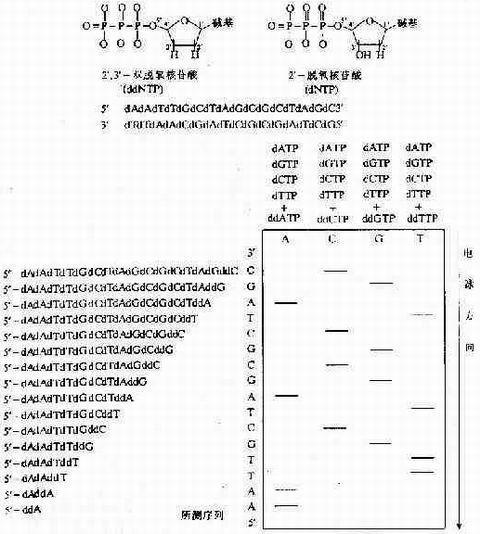
双脱氧
整个过程利用了DNA聚合酶的两个性质:(1)它能合成单链DNA模板的互补拷贝;(2)它能以2′,3′-双脱氧核苷三磷酸酯为底物。一旦当这一类似物被结合,其3′-端因缺少羟基而不再是链伸长所需的底物,从而使伸长中的DNA链终止。在实际操作中,使用DNA聚合酶I的克伦诺片段(Klenow fragment),它缺乏完整酶的5′,3′-核酸外切酶的活性。DNA的合成是在有四个脱氧核苷三磷酸酯的存在下进行,其中有一个或几个用32p作了标记,四个反应混合物中的每一个都含有一个双脱氧物。当反应结束时,最终反应混合物的每一个反应物中都有一系列新DNA的片段,都有一个相同的5′-端,但长度各不相同,并以碱基-特定3′-端为终止。这些混合物可用电泳法分离,用放射自显影术(射线显迹法)使之可视化,并读出序列。1980年桑格因设计出这种测定DNA(脱氧核糖核酸)内核苷酸排列顺序的方法而与W·吉尔伯特、P·伯格共获1980年诺贝尔化学奖。
原理通常DNA的复制需要:DNA聚合酶,单链DNA模板,带有3'-OH末端的单链寡核苷酸引物,4种dNTP(dATP、dGTP、dTTP和dCTP)。聚合酶用模板作指导,不断地将dNTP加到引物的3'-OH末端,使引物延伸,合成出新的互补DNA链。如果加入一种特殊核苷酸,双 脱氧核苷三磷酸(ddNTP),因为它与普通dNTP不同,在脱氧核糖的3’位置缺少一个羟基,故不能同后续的dNTP形成磷酸二酯键。例如,存在ddCTP、dCTP和三种其他的dNTP(其中一种为α-32P标记)的情况下,将引物、模板和DNA聚合酶一起保温,即可形成一种全部具有相同的5'-引物端和以ddC残基为3’端结尾的一系列长短不一片段的混合物。经变性聚丙烯酰胺凝胶电泳分离制得的放射性自显影区带图谱将为新合成的不同长度的DNA链中C的分布提供准确信息,从而将全部C的位置确定下来。采用类似的方法,在ddATP、ddGTP和ddTTP存在的条件下,可同时制得分别以ddA、ddG和ddT残基为3'端结尾的三组长短不一的片段。将制得的四组混合物全部平行地点加在变性聚丙烯酸受凝胶电泳板上进行电泳,每组制品中的各个组分将按其链长的不同得到分离,从而制得相应的放射性自显影图谱。从所得图谱即可直接读得DNA的碱基序列。
简介
现行的逻终止法人加减法序列测定技术(Sacger和Coulson,1975)发展而来的。加减法首次引入了使用特异引物在DNA聚合酶作用下进行延伸反应、碱基特异性的链终止,以及采用聚丙烯酰胺凝胶区分长度差一个核苷酸的单链DAN等3种方法。尽管有了这些进展,但加减法仍然太不精确,也太不得法,因此难以广为接受。直至引入双氧核苷三磷酸(ddTBP)作为链终止剂(Sanger等,1977 ),酶法DNA序列测定技术才得到广泛应用。2',3'ddNTP与普通dNTP不同之处在同它们在脱氧核糖的3' 位置缺少一个羟基。它们可以在DNA聚合酶作用下通过其5' 三磷酸基团掺入到正在增长的DNA链中,但由于没有3'羟基,它们不能同后续的dNTP形成磷酸二酯链,因此,正在增长的DNA链不可能继续延伸。这样,在DNA合成反应混合物的4种普通dNTP中加入少量的一种ddNTP后,链延伸将与偶然发生但却十分特异的链终止展开竞争,反应产物是一系列的核苷酸链,其长度取决于从用以起始DNA合成的引物末端到出现过早链终止的位置之间的距离。在4组独立的酶反应中分别采用4种不同的ddNTP,结果将产生4组寡核苷酸,它们将分别终止于模板链的每一个A、每一个G或每一个T的位置上。
Sanger法的试剂
引物酶促测序反应中利用一个与模板链特定序列互补的合成寡核苷酸作为DNA合成的引物。在许多情况下,可将靶DNA片段克隆于M13噬菌体或噬菌粒载体,以取得单链DNA分子作为模板。但也可以采用Sanger 法商定变性双链DNA模板的序列。在以上两种情况下,可以采用能与位于靶DNA侧翼的载体序列相退火的通用引物,而不必取得与未知DNA序列互补的引物。适M13噬菌体重组克隆的通用测序引物一般长15-29 个核苷酸,并可与紧靠M13mp18噬菌体多克隆位点区的HindⅢ位点成M13mp19 噬菌体多克隆位点区的EcoRI位点的序列互补。这些引物同样也可用于对克隆于pUC质粒的DNA进行“双链”测序,并可从许多厂商中购置得到。此外,还有若干家公司出售一些引物,这些引物下为了对通过多种限制酶切位点克隆于不同质粒的靶DNA进行测序而设计的。
模板如上所述,有两类DNA可以用作Sanger 法测序的模板:纯单链DNA和经过热变性或碱变性的双链DNA。采用通常从重组M13噬菌体颗粒中分离得到的单链DNA应中获得数百个核苷酸的序列。如用变性双链DNA作模板,则较难获得这一质量的结果,尽管采用双链DNA模板的方法显然既简单又方便(Chen和Seeburg,1985)。
然而只是在不久前得到改进以后,这一方法才发展到能够获得明确可信结果的水平。其中有两个因素是至关重要的,这就是模板DNA的质量和所用DNA聚合酶的种类。小量制箅的质粒DNA常常被寡脱氧核糖核苷酸小分子、核糖苷酸及DNA聚合酶的抑制剂所污染,其中前两种污染物可被用作随机引物。结果,种种“鬼”带、强终止现象,以及其他假象往往使测序凝胶含混不清、黯然失色。因此采用小量制备的质粒NDA来测定未知DNA克隆片段的序列,并不可取。然而,这类DNA常可作为对已经通过另一方法测定的序列进行进一步的合适模板。采用CsCl-溴化乙锭梯度平衡离心法来纯化质粒DNA,测序的结果会好得多,但却要耗费大量的人务和物力。模板链的每一个A、每一个G或每一个T的位置上。
DNA聚合酶
DNA聚合酶
通常用于双脱氧法序列测定的有几种不同的酶,其中包括大肠杆菌DNA聚合酶I的Klenow片段(Sanger等,1977),反转录酶(见文献,如Mieredorf和Prfeffer,1987)经过修饰消除了3'→5'外节酶活性的T7噬菌体DNA聚合酶(Sequenase)和测序酶2.0),Tabor和Richardson,1978]惟及从嗜热水生菌(T'hermus aquaticus)分离的耐热DNA聚合物(Taq DNA聚合酶)。这些酶的特性差别悬殊,因而可大大影响通过链终止反应所获得的DNA序列的数量的质量。大肠杆菌DNA聚合酶IKlenow 片段这种酶是最初用以建立Sanger法的酶,也是至今仍然广泛用于DNA序列测定的酶。通常碰到的两个问题是:
1)Koenow片段的持续合成能力低,以致一些片段并非由于dd NTP的掺入,而是因为聚合酸人模板上随机解离而终止合成,因而导致背景增高。由于该酶不能沿模板进行长中距离移动,因此利用该酶进行的标准测序反应所得序列的长度有限。通常,这一反应只能得到大约250-350个核苷酸的序列。如果分两步进行反应,所得序列的数目可以翻一番;其中第一步是初始标记步骤,采用低浓度的dNTP,而随后的第二步是链延伸-链终止反应,含有ddNTP和高浓度的dNTP(Johoston-Dow等,1987;Stambaugh和Blakesley,1988)。然而即使有了这些改进,用Klenow 酶所测序列的长度通常还是不如持续合成能力较强的测序酶。
2)这种酶对模板中的同聚核苷酸段或其他含牢固二级结构的区域进行复制的效能很低。将聚合反应的温度提高到55℃,可以缓解但并不能彻底解决这一问题(Gomer和Firtel,1985)。有时可采用一些dNTP类似物[如dITP或7-脱氮dGTP(7-deaza-dGTP)]来获取模板中可形成稳定二级结构的相应区段的序列信息,但Kleow酶对这些类似物的作用不如测序酶有效,这也许是因为它们使Klenow酶原已较低的持续合成能力进一步降低。
总而言这,可以选用大肠杆菌DNA聚合酶IKlenow片段测定从引物5'位置起250个碱基以内的一段DNA序列,但不宜用它来测定更长一段DNA序列或者具有二重对称和(或)同聚核苷酸段的DNA序列。
反转录酶尽管日常测序工作并不广泛使用反转录酶,但有时用这个酶解决一些由于模板DNA中存在A/T或G/C同聚核苷酸区而引起的问题。来自禽类和鼠类反转录病毒的反转录酶在这一看来要比Klenow酶略胜一筹(Karanthaansis,1982;Graham等,1986
),尽管它们也许还是比测序酶逊色(Cameron-Mills,1988;Revak等1988)。
测序酶测序酶(SequenaseTM)是一种经过化学修饰的T7噬菌体DNA聚合酶。这酶原来具有很强的3'→5'外切核酸活性,经过修饰后,这一活性大部分均被消除。测序酶2.0版是测序酶的基因工程产品,它完全缺失了3' →5'外切核酸酶活性,极其稳定而经活性要比经化学修饰的测序酶高2倍。测序酶持续合成能力很强,聚合速率很高,对诸如dITP和7-脱氮-dGTP等用于提高分子辨率使测序凝胶某些区段上的压缩条带得以分开的核苷酸类似物具有广泛的耐受性。它是测定长段DNA序列的首选酶。测序酶可以沿模板移动很长的距离,因而一套反应常常就可以测定数百个核苷酸的DNA序列。实际上,测得序列的长度更多是受聚丙烯酰胺凝胶的分辨能力而不是受该聚合酶的特性所制约。为了充分利用测序酶极高的持续合成能力,可采用两步测序反应。第一步首先采用低浓度的dNTP的较低温度,以便将合成反应限制在适度之下并确保放射性标记dNTP和较低温度,以便将合成反应限制在适度之下并确保放射性标记dNTP的有效掺入,这步反应的产物是仅仅延伸了20-30碱基的引物。再将第一步反应等分于4组1套的标准反应系统中,每组反应中都含有高浓度的dNTP和一种ddNTP。这样聚合反应就得以继续,直至造成链终止的核革酸掺入正在增长的链中。
Taq DNA聚合酶Taq DNA聚合酶适用于测定在37 ℃形成大段稳定十级结构的单链DNA模板序列。这是因为Taq
DNA聚合酶在70-75℃活性最高,这一温度下即使GC丰富的模板也无法形成二级结构。按照1nnis 等(1988)介绍的方法使用Taq DNA聚合酶进行测序,在放射自显影片上得到的测序梯连续数百个碱基条带始终清晰如一,表明这种酶的持续合成能力甚佳。模板链的每一个A、每一个G或每一个T的位置上。模板链的每一个A、每一个G或每一个T的位置上。
放射性标记的dNTP直至几年以前,实际上所有DNA测序反应都用[α-32P]dNTP来进行。然而32P发射的强β粒子造成两个问题。首先由于发生散射,放射自显影片上的条带远比凝胶上的DNA条带更宽、更为扩散,因此将影响到所读取的序列(尤其是从放射自显影片的上部所读取的序列)的正确性并将制约从单一凝胶上能读出的核苷酸序列的长度。其次32P的衰变会引起样品中DNA的辐射分解,因此用32P进行标记的测序反应只能保存一两天,否则DNA将被严重破坏以至测序凝胶上模糊不清、真假莫辨。[35S]dATP的引入(Biggin等,1983)大大缓解了上述两方面的矛盾。由于35S衰变产生较弱的β粒子,其散射有所减弱,凝胶和放射自显影片之间在分辨率上相差无几,因此可以从一套反应中确切测定数百核苷酸的DNA序列。此外,35S的低能辐射所引起的样品分解比较轻微,因此,测序反应可在-20℃保存至1周,而分辨率不见下降。这样,职果聚丙烯酰胺凝胶方面了发生技术故障,只要对测序反应进行重分析即可。
dNTP类似物
dNTP类似物
二重对称的DNA区段(特别是GC含量高者)可以形成链内二级过程中不能充分变性。因此将引起不规则迁移,使邻近的DNA条带压缩在一起,以致难以读出序列。这种压缩现象归因于DNA二级结构的存在,而且不可能通过改变测序反应中出序列。这种压缩现象归因于DNA二级结构地存在,而且不可能通过改变测序反应中所用DNA聚合酶的种类而得到减轻。但是凝胶中的压缩区段往往可以通过采用诸如dITP(2'-脱氧次黄苷15' -三磷酸)或7-脱氮-dGTP(7-脱氮-2'-脱氧鸟苷-5' -三磷酸)等核苷酸类似物进行分辨。这些类似物与普通碱基的配对能力较弱,而且是测序酶和Taq DNA聚合酶等DNA聚合酶的合适底物(Gough和Murray,1983;Mixusawa等,1986;Innis等,1988)。但对某些压缩条带,7-脱氮-dGTP无济于事;同样,dITP也无补于另一压缩条带(尤其是得于GC丰富区的缩条带)的分辨。如果需要采用类似物,首先可试用dOTP,如果压缩条带用d ITP或7-脱氮-dGTP都无法分辨,则转而测定另一条链的DNA序列几乎总能如愿以偿。如上所述,两种形式的测序酶和Taq DNA聚合酶对核苷酸类似物的耐受性优于大肠杆菌DNA聚合酶IKlenow片段。此外,制造厂商声称在测定含稳固二结构的模板序列时,测序酶2.0版要优于原来的测序酶。测序酶2.0版持续合成能力强于测序酶,其作用总是一气呵成,很少半途而废,因而消除了“鬼”带。而且,测序酶2.0版对诸如dITP类核苷酸类惟物的耐受性看来也优于原来的测序酶。测定战略
由于DNA一般都由几千个单核苷组成。而目前测定DNA序列的最好方法,一次也只能测约600个核苷酸,因此进行DNA顺序测定前,需要用不同限制酶消化等测DNA,使其降成小片段,分别克隆到pUC1,18pUCl18,pUC19,M13mp18,M13mp19等载体中,分别测定各小片段的顺序,由于不同限制酶产生的片段之间有交错重叠顺序,根据片段间和末端重叠序列,用计算机软件如Mac Vector TM5.0分析DNA,Assemblylign TM排列出各片段的位置,进而排出DNA的全序列。
测定方法
以M13噬菌体为载体的双氧链终止法的实施步骤为例:
M13噬菌体复制型双链DNA的制备M13噬菌体复制型双链DNA(RFDNA)的制备。
为了将待测双链DNA进行克隆,必须制备M13mp18或M13 mp19复制型(闭环双链)DNA,从M9培养基中,挑起单个克隆大肠杆菌,JM101到50ml2×YT培养基中,37℃振荡过夜。稀释1ml培养物到50ml2×YT培养中,于37℃振摇6h,转移2ml培养物于微量离心管中,12000g,离心5min细菌沉淀保留用于分离复制型RFDNA,上清用于分离噬菌体单链DNA。
细菌沉淀用于分离复制型DNA,可以采用标准的碱解方法或采用天美公司的Wizard Miniperps DNA试剂盒制备。(方法同分离质粒DNA方法)。
重组噬菌体制备采用多克隆位点上的限制性内加酶切割待测DNA,并采用相同内切酶切割M13噬菌体RFDNA(采用Biol 101gene clean Ⅱ试剂盒分别纯化内切酶切割后的DNA片段),然后将待测外源DNA片段与M13复制型载体DNA连接过夜,连接反应物转染JM101感受态细菌,将连接物10μl与200μl感受态细菌混匀,放置冰上40~50min,再放在42℃水浴2min,然后加入1ml新鲜的静止相JM101培养物混匀,然后分别以300μl于含有IPIG(200mg/ml)和X-gal200mg/ml的2×YT培养基上,于37℃培养8h即可出现蓝色和白色噬斑,白色或称无菌噬斑,即为阳性重组噬菌体。
单链噬菌体DNA(模板DNA)的制备上述平板的每个白色噬菌斑是由一种重组M13DNA分子转染细菌后产生的,如果取一个白色噬菌斑进行培养便可得到一种单链形式的DNA。为此,挑取一个白色噬菌斑转接到一个新鲜制备的OD600=0.1的大肠杆菌JM101培养物中,于37℃振摇5h左右,12000g离心10min,上清转移到另一新微量离心管中用于分离单链DNA,按1ml上清液中加入含20%聚乙二醇PEG-6000的2.5m NaCl 200μl沉淀噬菌体,再用饱和酚抽提除去噬菌体蛋白,最后用1/10体积的3m NaAc和乙醇沉淀单链DNA,浓度调至0.1~0.5μg/μl,也可采用天美公司的Wizard TM M13DNA纯化试剂盒分离纯化M13单链DNA。
引物制备可以购买通用引物或实验室合成15bp~26bp长度的引物,通常以2μg/ml的浓度贮存于-20℃备用。
微量滴板进行大批量的模板测序时,常在密闭的微量离心管中进行与引物的退火反应,然后在微量滴板中进行链延伸-链终止反应。
变性聚丙烯酰胺凝胶测序凝胶装置的大小形状均不相同,其主要参数有:①长度通常为40~50cm;②宽度通常为20cm;③厚度通常为0.3~0.4mm;④横截面形状为楔形或锥形,即顶部薄,底部厚,或者是将底部凝胶缓冲液的浓度提高;⑤加样槽;⑥整块凝胶上的温度应均一。
电泳后将凝胶干燥,放射自显影如标题
凝胶上读取DNA序列此时读取的是目的DNA的互补链3'~5'的序列。